
Desenredando
la complejidad del riesgo de diabetes: un enfoque bayesiano para el aprendizaje
de estructuras causales
Untangling the
complexity of diabetes risk: a Bayesian approach to
learning causal structures
Ney Michel Lituma Villamar 1,a
1. Universidad
de Guayaquil, Guayaquil, Ecuador.
a. Magíster
en Inteligencia Artificial Aplicada.
ORCID:
https://orcid.org/0000-0002-2820-6655
Citar como: Lituma-Villamar NM. Desenredando la complejidad del riesgo
de diabetes: un enfoque bayesiano para el aprendizaje de estructuras causales. Rev Perú Cienc Salud. 2025;7(3):#-#. doi: xxxx
RESUMEN
Objetivo. Evaluar el rendimiento e interpretabilidad
de clasificadores de redes bayesianas para la detección temprana de diabetes. Métodos. Se realizó un estudio de
validación de modelos de aprendizaje automático (machine learning) aplicado al campo de la
salud, enfocado en la evaluación de rendimiento y explicabilidad
de algoritmos sobre un conjunto de datos categóricos y preprocesado.
Específicamente, fueron entrenados y aplicados: Naive
Bayes, Tree Augmented Naive-Chow-Liu (TAN–Chow-Liu), Tree Augmented Naive-Hill Climbing with Super
Parents (TAN–HCSP), Fast Super-Parent Search with Joint Mutual Information (FSSJ) y K-Dependence
Bayesian Classifier (KDB),
sobre 100 000 registros preprocesados (filtrados por
su relevancia causal y discretización de variables)
utilizando bnlearn
y bnclassify.
La partición fue 75/25 (entrenamiento/prueba) y fueron estimadas exactitud,
sensibilidad, especificidad y F1; además, fueron analizadas las estructuras
aprendidas frente a la evidencia clínica. Resultados.
Palabras
clave: diabetes mellitus; redes bayesianas; inteligencia
artificial; índice de masa corporal; hipertensión; hemoglobina a glucosilada; algoritmos; factores de riesgo; pronóstico;
diagnóstico precoz (fuente: DeCS-BIREME).
ABSTRACT
Objective: To
evaluate the performance and interpretability of Bayesian network classifiers
for the early detection of diabetes. Methods:
A model validation study of machine learning applied to healthcare was
conducted, focusing on performance assessment and explainability
of algorithms on a categorical and preprocessed dataset. Specifically, the
following classifiers were trained and applied: Naive Bayes, Tree Augmented
Naive–Chow-Liu (TAN–Chow-Liu), Tree Augmented Naive–Hill Climbing with Super
Parents (TAN–HCSP), Fast Super-Parent Search with Joint Mutual Information
(FSSJ), and the K-Dependence Bayesian Classifier (KDB). Models were tested on
100,000 preprocessed records (filtered by causal relevance and variable
discretization) using bnlearn and bnclassify. Data were partitioned
75/25 (training/testing), and accuracy, sensitivity, specificity, and F1 score
were estimated. In addition, the learned structures were analyzed against
clinical evidence. Results:
Keywords: diabetes
mellitus; Bayesian networks; artificial intelligence; body mass index;
hypertension; glycated hemoglobin A; algorithms; risk factors; prognosis; early
diagnosis (source: MeSH-NLM).
INTRODUCCIÓN
La
detección temprana de la diabetes es crítica para reducir morbilidad y
mortalidad. A escala global, más de 422 millones de personas viven con la
enfermedad y se atribuyen 1,5 millones de muertes anuales, con mayor carga en
países de ingresos bajos y medianos (1). En 2021, la Federación
Internacional de Diabetes (IDF) (2) estimó que padecían
diabetes 537 millones de adultos (10,5 % de la población); lo que generó un
gasto sanitario de 966 mil millones de dólares, con proyecciones de 643
millones en 2030 y 783 millones en 2045, superando 1054 mil millones en costos.
Por
lo expuesto, la personalización terapéutica requiere hipótesis causales
sólidas, ya que el descubrimiento causal basado en datos puede apoyar la toma
de decisiones clínicas (3). Diversos algoritmos de aprendizaje
automático superan el 85 % de precisión en la predicción de diabetes (4)
y detectan patrones sutiles en enfermedades raras (5). En este
contexto, las redes bayesianas (BN) destacan por manejar incertidumbre y
ofrecer interpretabilidad (6,8), además de
su flexibilidad para variables categóricas y continuas, la integración de conocimiento
experto y la inferencia con datos faltantes (9,11). La evidencia
previa en diabetes reportó valores predictivos positivos y negativos de 69,6 %
y 79,9 % en mujeres (12), así como dependencias entre atributos
relevantes usando enfoques bayesianos (13). Un reto persistente es
el desequilibrio de clases, que reduce el rendimiento de los clasificadores (14);
para mitigarlo, fueron aplicadas las técnicas de sobremuestreo
sintético de minorías (SMOTE) (15), submuestreo
(16), estrategias híbridas (17), selección ponderada de
características con Random Forest
y XGBoost (18), y aprendizaje sensible al
costo con reducción de dimensionalidad (19).
En BN, la combinación de detección de características y remuestreo
múltiple ha facilitado la identificación de factores de riesgo (20),
y es clave distinguir una BN generativa de un clasificador BN optimizado para
precisión (21). Estudios recientes amplían el marco: integración de
BN para diabetes tipo 2 (T2D) y enfermedades coronarias (CHD) (22),
uso de mantas de Markov en población con prediabetes (23)
y exploración de interacciones de biomarcadores con
búsqueda Tabú (Tabu search) y bootstrap (24).
Pese
al avance metodológico, persisten lagunas: a) faltan comparaciones cabeza a
cabeza de múltiples clasificadores bayesianos bajo un preprocesamiento
estandarizado en grandes bases categóricas con desequilibrio; b) no está
claramente delimitado cuándo arquitecturas básicas ⎯por ejemplo, Naive Bayes o K-Dependence Bayesian Classifier (KDB)⎯ igualan o superan a variantes más complejas, como las
Tree Augmented Naive Bayes (TAN), ya sea en sus
variantes Chow-Liu, Hill Climbing
with Statistical Perturbation (HCSP) o Forward Sequential
Selection and Joining
(FSSJ), en términos de utilidad clínica; y c) se necesita verificar que las
estructuras aprendidas sean plausibles clínicamente y destaquen factores
modificables para su intervención. Abordar estas brechas es relevante para
sistemas de apoyo a la decisión y para priorizar estrategias de prevención y manejo
del riesgo.
El
objetivo de este estudio fue comparar el rendimiento e interpretabilidad
de clasificadores bayesianos Naive Bayes, Tree Augmented
Naive-Chow-Liu (TAN–Chow-Liu),
Tree Augmented Naive-Hill Climbing with Super Parents
(TAN–HCSP), Fast Super-Parent
Search with Joint Mutual Information (FSSJ) y
K-Dependence Bayesian Classifier KDB) aplicados a un conjunto de 100 000
registros de predictores de diabetes (25), tras preprocesamiento
(discretización y filtrado por relevancia causal) y
estrategias de manejo del desequilibrio, evaluando exactitud, sensibilidad,
especificidad y puntuaciones F1, así como contrastando las estructuras
aprendidas con la literatura clínica existente.
MÉTODOS
Se realizó un estudio de validación de modelos de
aprendizaje automático (machine learning) aplicado al campo de la salud, enfocado en la
evaluación de rendimiento y explicabilidad de
algoritmos sobre un conjunto de datos categórico y preprocesado.
Se utilizó el Diabetes Health Indicators
Dataset, un conjunto de datos público disponible en Kaggle con 100 000 registros y 16 predictores de diabetes (25),
derivado de las encuestas del Behavioral Risk Factor Surveillance System (BRFSS) de los Centers for
Disease Control and Prevention
(CDC) de EE. UU. La elección de este dataset se justifica por su gran tamaño muestral
mayor a 250 000 registros, que proporciona la potencia estadística necesaria
para el entrenamiento y la validación de los modelos de aprendizaje automático.
Su accesibilidad y estructura lo convierten en un recurso valioso para
investigaciones exploratorias sobre la viabilidad de algoritmos de inteligencia
artificial (IA) en la identificación de factores de riesgo en grandes
poblaciones, facilitando la reproducibilidad y la comparación de resultados en
la comunidad científica (26).
En
la preparación, no se detectaron valores perdidos; las cinco columnas de raza
se unificaron en una variable categórica, mientras que la edad y el IMC fueron discretizados; año y región se excluyeron por baja
relevancia causal. Todos los campos se transformaron a factores en el programa
R y el conjunto se barajó y dividió en un 75 % sobre un 25 %
(entrenamiento/prueba) con reproducibilidad garantizada. Se entrenaron Naive Bayes, TAN–Chow-Liu, TAN–HCSP, FSSJ y KDB con bnclassify (suavizado de Laplace)
y posteriormente se refinaron con bnlearn. Conceptualmente, Naive Bayes asumió independencia condicional; TAN incorporó un
árbol de dependencias; TAN–HCSP añadió restricciones jerárquicas; FSSJ realizó
selección progresiva de variables; y KDB combinó k-vecinos con RB dinámicas
para datos con deriva temporal. Se obtuvieron exactitud, precisión, recall y F1
superiores a 0,90 en todos los clasificadores; TAN–Chow-Liu
proporcionó el mapa de dependencias más claro y FSSJ mostró la mayor eficiencia
computacional. Las estructuras aprendidas se inspeccionaron con gRain y se visualizaron mediante Rgraphviz,
confirmándose enlaces clínicamente plausibles entre predictores clave y
diabetes.
RESULTADOS
Con
el paquete bnlearn
fueron generados tres grafos acíclicos dirigidos
(DAG) que hicieron explícitas las estructuras bayesianas aprendidas (ver
Figuras 1, 2 y 3). Con estas visualizaciones fue desplazado el foco desde la
exactitud predictiva bruta hacia las dependencias probabilísticas subyacentes,
permitiendo que sea evaluado cómo es codificado el conocimiento clínico por
cada algoritmo (ver Tabla 1).
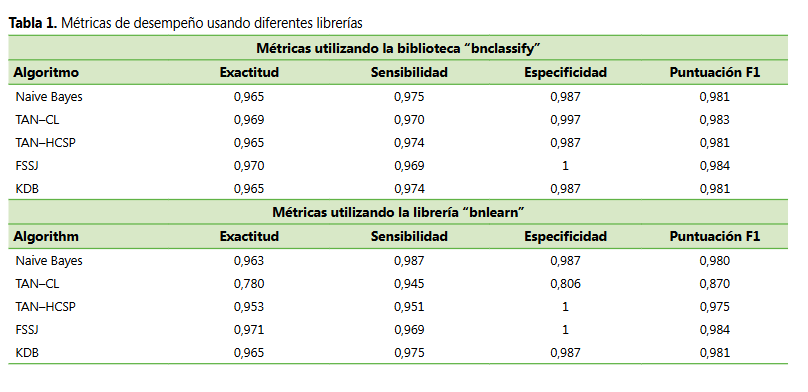
En
la Figura 1 queda representada la topología en estrella de Naive
Bayes: cada predictor es modelado como hijo directo
de la diabetes, sin enlaces laterales entre las variables. Se muestra un nivel
adicional en el que la edad canaliza el riesgo hacia la hipertensión,
enfermedad cardíaca e IMC, destacándose la edad como el conducto por el cual la
diabetes “explica” la morbilidad subsecuente. La suposición de independencia
simplifica el cómputo, pero aplana correlaciones del mundo real; por ejemplo,
glucosa y hemoglobina glicosilada (HbA1c) no
interactúan entre sí, aun cuando se encuentran fisiológicamente
interrelacionadas.
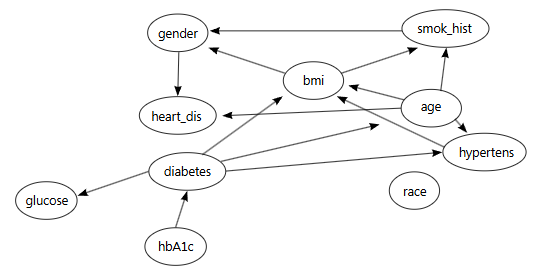
Figura 1. Gráfico del modelo bayesiano de Naive
Bayes
Con
el aprendiz TAN–Chow–Liu
fue generado un árbol que situó al IMC en la raíz, desde la cual se irradiaron
tres trayectorias epidemiológicas: historia de tabaquismo, IMC y sexo,
capturándose patrones de consumo específicos por sexo, edad, IMC e hipertensión,
lo que refleja el aumento de la presión arterial mediado por el peso y el
envejecimiento; así mismo, el IMC hacia la enfermedad cardíaca y, finalmente,
la diabetes, considerada una cascada cardio-metabólica
canónica. La glucemia sostenida (HbA1c) fue modelada como entrada directa a la
diabetes, mientras que la glucosa y la raza/etnia permanecieron aisladas, lo
que sugiere que, en esta cohorte, la exposición crónica constituye una señal
más robusta que una medición aislada de glucosa y que la variabilidad étnica
sería negligible (ver Figura 2).
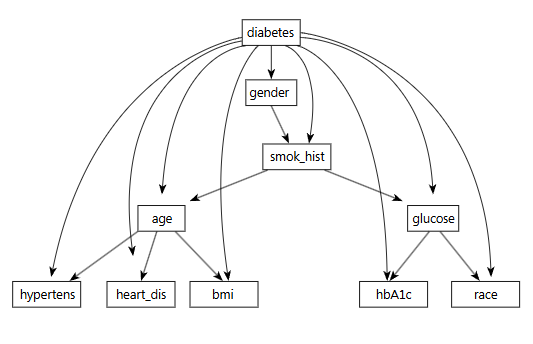
Figura 2. Gráfico del modelo bayesiano de Chow-Liu
La
Figura 3 fue generada por el algoritmo TAN–HCSP, enriqueciéndose el árbol sin
perder interpretabilidad. La HbA1c fue modelada como
“superpadre” de la diabetes, la cual, a su vez, se
encadenó hacia la edad y, a través de la edad, hacia el IMC y la historia de
tabaquismo, alcanzándose finalmente el sexo y la enfermedad cardíaca. Fue
cerrado el circuito metabólico con el bucle IMC asociado con la hipertensión y
esta, a su vez, con la diabetes; así mismo, la glucosa se conectó directamente
con la diabetes. Con esta arquitectura quedaron capturadas cadenas de múltiples
pasos: cómo la edad influye en el IMC, y este, a su vez, en la glucosa y,
finalmente, en la diabetes, conciliándose así la simplicidad con un mayor
detalle epidemiológico.
Figura 3. Gráfico del modelo bayesiano TAN–HCSP (Hill-Climbing)
DISCUSIÓN
Los factores de riesgo identificados por nuestro
modelo, como un alto IMC, inactividad física e hipertensión, son consistentes
con las guías de práctica clínica de la Asociación Americana de Diabetes (ADA)
y la Federación Internacional de Diabetes (IDF), lo que valida su capacidad
para reconocer patrones clínicamente relevantes (27,28). Sin
embargo, a diferencia de los umbrales de riesgo fijos de las guías, el modelo
de IA ofrece una visión más granular, al descubrir interacciones complejas y no
lineales entre variables, lo que podría complementar y personalizar la
estratificación del riesgo en el futuro (29). Es crucial interpretar
estos hallazgos como asociaciones y no como causalidad directa, dado el diseño
transversal del estudio.
Se
observó una trayectoria directa desde niveles elevados de hemoglobina glucosilada (HbA1c) hacia la presencia de diabetes, lo que
respalda el papel de la hiperglucemia crónica como eje de progresión, desde
estados intermedios a enfermedad manifiesta, así como su valor como marcador
diagnóstico y pronóstico en la práctica clínica (23,30,31). Además,
aun en ausencia de diagnóstico, valores altos de HbA1c se han asociado con
mayor incidencia de eventos cardiovasculares a mediano plazo, lo que refuerza
su relevancia clínica más allá del umbral diagnóstico (31).
Por
otro lado, la trayectoria el IMC, la hipertensión y enfermedad cardíaca
identificada por la red fue congruente con la evidencia que vincula la
adiposidad con el aumento del riesgo de hipertensión y, subsecuentemente, de
enfermedad cardiovascular (32,33). Desde cierto punto de vista, la
obesidad se ha relacionado con la resistencia a la insulina, la disfunción
endotelial y la inflamación crónica, es decir, elementos que facilitan el
desarrollo de la hipertensión y la aterosclerosis. Se ha informado, además, que
la hipertensión puede actuar como mediador principal de las secuelas
cardiovasculares de la obesidad (33). En consecuencia, el control
del peso fue considerado una estrategia cardinal para interrumpir dicha cadena
causal y reducir la carga de la enfermedad cardiovascular (32,33).
El
contraste con la literatura reciente, este estudio mostró concordancia con
modelos bayesianos aplicados en prediabetes, en los que la HbA1c y el IMC
emergieron como factores de mayor influencia en la progresión hacia la diabetes
tipo 2 y en la estratificación de riesgo (23). Del mismo modo, la
coexistencia de alteraciones glucémicas y factores cardio-metabólicos
se asoció con un incremento relevante del riesgo cardiovascular, lo que subraya
la necesidad de valorar el riesgo de manera integral y no aislada (34,35).
En conjunto, las trayectorias inferidas sugieren que la intervención clínica
temprana sobre el control glucémico y el peso corporal podría traducirse en
beneficios, tanto metabólicos como cardiovasculares (31,33,35).
Una
contribución práctica de este enfoque radicó en la capacidad de las redes
bayesianas para representar y comunicar interdependencias clínicas entre
variables, así como para responder a preguntas contrafácticas
(“¿qué ocurriría si…?”) de utilidad en la toma de decisiones; ello permitió
visualizar rutas directas e indirectas (p. ej., IMC, hipertensión y
cardiopatía) o priorizar intervenciones de acuerdo con su impacto potencial
sobre el riesgo individual (30).
Entre
las limitaciones del estudio se encuentra el riesgo de sesgo poblacional, ya
que el dataset
derivado del BRFSS refleja principalmente la demografía de Estados Unidos y
puede subrepresentar a grupos étnicos con perfiles de
riesgo distintos (36). Este sesgo compromete la validez externa del
modelo, pues un algoritmo entrenado en una población específica puede mostrar
un rendimiento deficiente y no generalizable en otros contextos demográficos,
llegando incluso a exacerbar las disparidades en salud existentes, como ha sido
documentado ampliamente en la investigación de IA y salud (37,38).
Por lo tanto, antes de considerar una implementación clínica es imperativo que
futuras investigaciones validen prospectivamente el modelo en cohortes locales
y diversas, empleando conjuntos de datos más inclusivos y metodologías de
equidad en IA (fairness-aware machine learning)
para asegurar que sus beneficios sean universales y no se limiten a la
población original del estudio (39).
Otro
aspecto fundamental a considerar es que la integración de herramientas de IA en
la predicción de diabetes conlleva importantes implicaciones éticas y
regulatorias que requieren un abordaje proactivo. Entre estas destaca la
problemática de la "caja negra", inherente a algoritmos complejos, la
cual compromete tanto la autonomía del paciente como la responsabilidad
profesional del clínico, evidenciando la necesidad imperativa de desarrollar
sistemas de IA explicable (XAI) que faciliten la toma de decisiones médicas
fundamentadas y transparentes (40). Desde la perspectiva regulatoria,
agencias como la FDA y EMA están desarrollando marcos para la aprobación de software como dispositivo médico (SaMD), que exigen una validación clínica rigurosa,
monitorización continua posimplementación y
protocolos de privacidad conforme a normativas como HIPAA o RGPD (41,42).
Finalmente, es imperativo garantizar equidad en el acceso a estas tecnologías
para evitar ampliar brechas digitales y sanitarias, asegurando que los
algoritmos de estratificación de riesgo diabético sean herramientas para
intervenciones preventivas universalmente accesibles y no fuentes de
discriminación en atención médica o seguros de salud (39).
Como
agenda integrada de mejora, se sugiere: a) enriquecimiento de variables y
estructura: incorporar conexiones explícitas para sexo y raza/etnia por su
impacto en riesgo diabético y cardiovascular; ampliar la caracterización de
tabaquismo (activo/pasivo) por sus efectos indirectos vía salud vascular;
añadir hábitos dietéticos, actividad física e historia familiar (hoy ausentes o
poco granulares); extender biomarcadores (perfil
lipídico, función renal, marcadores inflamatorios) para capturar vías
fisiopatológicas adicionales; e integrar factores genéticos y ambientales (p.
ej., puntajes poligénicos, exposición a
contaminantes) para una evaluación más completa del riesgo; b) diseño y
validación rigurosa: ejecutar estudios longitudinales multicéntricos
que aseguren precedencia temporal y validez externa; validar por subgrupos
(edad, sexo, etnia, comorbilidades) para auditar equidad; reportar calibración
(Brier score, curvas de calibración) y
utilidad clínica (curvas de decisión) además de discriminación; y abordar el
desbalance con estrategias de muestreo y aprendizaje sensible al costo
cuantificando su efecto en sensibilidad, especificidad y equidad; c)
intervenciones y traslado: diseñar y
probar intervenciones dirigidas a cadenas indirectas (p. ej., IMC, hipertensión
y cardiopatía), priorizando factores modificables (IMC, presión arterial);
emplear inferencia contrafáctica para estimar el
impacto de pérdidas de peso del 5-10 % o intensificación antihipertensiva sobre
riesgo futuro de diabetes y eventos CV; e integrar datos dinámicos (MCG,
presión domiciliaria, wearables) y
modelos temporales (DBN/KDB) para capturar deriva y trayectorias; d)
implementación y gobernanza: integrar progresivamente en la historia clínica
electrónica (HCE) con clínica
explicable (rutas causales, contribuciones por variable), salvaguardas éticas y
auditorías periódicas; establecer ciclos de actualización (reentrenamiento y
revisión de umbrales) con monitoreo de desempeño, equidad y seguridad; y
definir protocolos de uso (quién/cuándo/cómo) apoyados con materiales
educativos para asegurar adopción segura y efectiva.
CONCLUSIONES
A
pesar de sus diferencias estructurales, todos los clasificadores superaron una
exactitud de 0,96 tras la validación cruzada. El FSSJ encabezó con una
aproximación de 0,97 de exactitud y especificidad perfecta, mientras que Naive Bayes, TAN–HCSP y KDB
ofrecieron un desempeño alto y estable. El TAN–CL resultó más sensible a las
idiosincrasias de los datos, aunque permitió identificar nuevas rutas causales.
Estos resultados confirman que, cuando las relaciones entre variables son
sencillas, las arquitecturas bayesianas básicas pueden igualar a variantes más
elaboradas. Aun así, las herramientas de aprendizaje de estructura, como bnlearn, siguen siendo indispensables para
extraer conexiones clínicamente interpretables, aportando conocimientos que
pueden orientar estrategias de prevención focalizadas más allá de lo que
comunican las métricas escalares de desempeño.
Por
otro lado, se concluye que los resultados se consideran útiles para la
estratificación clínica del riesgo cardio-metabólico
y para la toma de decisiones personalizadas. La representación causal obtenida
permitiría priorizar intervenciones sobre factores modificables (p. ej.,
control ponderal y presión arterial) y comunicar riesgos en términos
comprensibles para pacientes y equipos clínicos.
En
este sentido, el estudio demuestra que la complejidad arquitectural de los
clasificadores bayesianos no se traduce necesariamente en mejoras predictivas
proporcionales, dado que todos los modelos evaluados alcanzaron un rendimiento
comparable. Los hallazgos subrayan la importancia de equilibrar precisión
predictiva con interpretabilidad clínica,
posicionando estas metodologías como herramientas valiosas para la medicina de
precisión. La capacidad de generar representaciones causalmente interpretables
facilita tanto la estratificación de pacientes como el desarrollo de
estrategias preventivas personalizadas, lo que representa un avance
significativo hacia la optimización de los resultados en salud cardiovascular
mediante intervenciones dirigidas a factores de riesgo modificables.
REFERENCIAS BIBLIOGRÁFICAS
- Organización
Mundial de Salud. Informe Mundial de la Diabetes [Internet]. Ginebra: OMS;
10 de septiembre de 2024 [Consultado 9 de julio de 2025]. Disponible en: https://iris.who.int/bitstream/handle/10665/254649/9789243565255-spa.pdf
- Hossain J, Al-Mamun,
Islam R. Diabetes mellitus, the fastest growing global public health
concern: Early detection should be focused. Health Sci Rep. [Internet].
22 de marzo de 2024 [Consultado el 9 de julio de 2025];7(3):e2004. doi: 10.1002/hsr2.2004
- Bronstein M, Meyer-Kalos
P, Vinogradov S, Kummerfeld
E. Causal Discovery Analysis: A Promising Tool for Precision Medicine. Psychiatr Ann. [Internet]. 2024 [Consultado el 9 de julio
de 2025];54(4):e119-e124. https://doi.org/10.3928/00485713-20240308-01
- Montero Rodríguez JC de J, Roshan
Biswal R, Sánchez de la Cruz E. Algoritmos de
aprendizaje automático de vanguardia para el diagnóstico de enfermedades.
Res Comput Sci.
[Internet]. 2019 [Consultado el 9 de julio de 2025];148(7):455-68.
Disponible en: https://rcs.cic.ipn.mx/2019_148_7/Algoritmos%20de%20aprendizaje%20automatico%20de%20vanguardia%20para%20el%20diagnostico%20de%20enfermedades.pdf
- Gómez Ruiz I. Diseño e implementación de modelos
de lenguaje para información genómica asociada a enfermedades raras
mediante inferencia gramatical [Internet]. Valencia: Universitat
Politècnica de València;
2024 [Consultado el 9 de julio de 2025]. Disponible en: https://riunet.upv.es/server/api/core/bitstreams/e752670f-3702-4eee-846b-c16237a5f925/content
- Darwiche A. Modeling and Reasoning with Bayesian Networks
[Internet]. Cambridge:
Cambridge University Press;
2009 [Consultado el 9 de julio de 2025]. Disponible en: https://books.google.co.ve/books?id=7AjXGltje7YC&printsec=frontcover#v=onepage&q&f=false
- Hassija V, Chamola V, Mahapatra A, Singal A, Goel D, Huang K, et al. Interpreting Black-Box Models:
A Review on Explainable Artificial Intelligence. Cogn Comput. [Internet].
2024 [Consultado el 9 de julio de 2025];16:45-74.
doi: 10.1007/s12559-023-10179-8
- Lucas PJ, Van der Gaag
LC, Abu-Hanna A. Bayesian networks in biomedicine and health-care. Artif Intell Med. [Internet]. 2004 [Consultado el 9 de julio de
2025];30(3):201-14. doi: 10.1016/j.artmed.2003.11.001
- Koller D, Friedman N. Probabilistic Graphical Models:
Principles and Techniques. [Internet].
Cambridge: MIT Press; 2009 [Consultado el 9 de
julio de 2025]. Disponible en: https://www.researchgate.net/publication/220690050_Probabilistic_Graphical_Models_Principles_and_Techniques
- Pearl
J. Causality: Models, Reasoning and Inference. [Internet]. 2ª ed. Cambridge: Cambridge University Press; 2009
[Consultado el 9 de julio de 2025]. Disponible en: https://dl.acm.org/doi/book/10.5555/1642718
- Suo X, Huang X, Zhong L,
Luo Q, Ding L, Xue F. Development and Validation
of a Bayesian Network‐Based Model
for Predicting Coronary Heart Disease Risk From
Electronic Health Records. J
Am Heart Assoc. [Internet].
2 de junio de 2024 [Consultado el 9 de julio de 2025];13(1):e029400. doi: 10.1161/JAHA.123.029400
- Coaquira-Flores EE, Torres-Cruz F, Condori-Quispe SJ, Tisnado-Puma JC, Melgarejo-Bolivar
RP, Herrera-Urtiaga AP, et al. Predicción de
diabetes en mujeres mediante un modelo probabilístico basado en redes
bayesianas. Científica Digit. [Internet]. 29 de
abril de 2023 [Consultado el 9 de julio de 2025];16:185-201.
doi: 10.37885/230412748
- Bressan GM, Flamia de Azevedo
BC, Molina de Souza R. Métodos de classificação automática para predição
do perfil clínico de pacientes portadores do diabetes mellitus. Braz J Biometrics.
[Internet]. 29 de junio de 2020 [Consultado el 9 de julio de 2025];38(2):257-73.
https://doi.org/10.28951/rbb.v38i2.445
- Ndjaboue R, Ngueta G, Rochefort-Brihay C, Delorme S, Guay
D, Ivers N, et al. Prediction models of diabetes
complications: a scoping review. J Epidemiol Community
Health [Internet]. 30 de junio de 2022
[Consultado el 9 de julio de 2025];76(10):896-904. doi:
10.1136/jech-2021-217793
- Alghamdi M, Al-Mallah M, Keteyian S, Brawner C, Ehrman C, Sakr S. Predicting
diabetes mellitus using SMOTE and ensemble machine learning approach: The
Henry Ford ExercIse Testing (FIT) project. PLoS One [Internet]. 2017
[Consultado el 9 de julio de 2025];12(7):e0179805.
doi: 10.1371/journal.pone.0179805
- Nejatian S, Parvin H, Faraji E. Using sub-sampling and ensemble clustering
techniques to improve performance of imbalanced classification. Neurocomputing [Internet]. 7 de febrero de 2018 [Consultado el
9 de julio de 2025];276:55-66. https://doi.org/10.1016/j.neucom.2017.06.082
- Praveenkumar KS. Un enfoque híbrido de analítica de big data para predecir diabetes tipo II usando H-SMOTE
tree. Adv Nanotechnol Mater Sci Eng Innov. [Internet]. 2024
[Consultado el 9 de julio de 2025];20(S2):606-624. https://doi.org/10.62441/nano-ntp.vi.494
- Xu Z, Wang Z. A Risk Prediction Model for Type 2
Diabetes Based on Weighted Feature Selection of Random Forest and XGBoost Ensemble Classifier. In: 11th Int Conf Adv
Comput Intelligence (ICACI); Guilin, China 2019
Jun 7-9. [Internet]. Guilin, China: Instituto de Ingenieros Eléctricos y
Electrónicos: 2019: 278-283 [Consultado el 9 de julio de 2025].
doi:10.1109/ICACI.2019.8778622
- Pes B, Lai G. Cost-sensitive learning strategies
for high-dimensional and imbalanced data: a comparative study. PeerJ Comput Sci. [Internet]. 2021 [Consultado el 9 de julio de
2025];7:e832. doi: 10.7717/peerj-cs.832
- Wang X, Ren J, Ren H, Song W, Qiao
Y, Zhao Y, et al. Diabetes mellitus early warning and factor analysis
using ensemble Bayesian networks with SMOTE-ENN and Boruta.
Sci Rep. [Internet]. 2023 [Consultado el 9 de julio
de 2025];13:12718. doi:
10.1038/s41598-023-40036-5
- Parrales-Bravo F, Caicedo-Quiroz
R, Rodríguez-Larraburu E, Barzola-Monteses
J. ACME: A Classification Model for Explaining the Risk of Preeclampsia
Based on Bayesian Network Classifiers and a Non-Redundant Feature
Selection Approach. Informatics [Internet].
2024 [Consultado el 9 de julio de 2025];11(2):31. https://doi.org/10.3390/informatics11020031
- Kong D, Chen R, Chen Y, Zhao L, Huang R, Luo L,
et al. Bayesian network analysis of factors influencing type 2 diabetes,
coronary heart disease, and their comorbidities. BMC Public Health. [Internet]. 8 de mayo de 2024 [Consultado el 9
de julio de 2025];24:1267. doi:
10.1186/s12889-024-18737-x
- Fuster-Parra P, Yañez AM, López-González A, Aguiló A, Bennasar-Veny M. Identifying risk factors of
developing type 2 diabetes from an adult population with initial
prediabetes using a Bayesian network. Front Public Health. [Internet]. 2023 [Consultado el 9 de julio de
2025];10:1035025. doi: 10.3389/fpubh.2022.1035025
- Sun Y, Lei J, Kosmas P. Exploring Biomarker
Relationships in Both Type 1 and Type 2 Diabetes Mellitus Through a
Bayesian Network Analysis Approach. arXiv
[Preprint]. 2024;
arXiv:2406.17090. https://doi.org/10.48550/arXiv.2406.17090
- Choksi P. Conjunto de datos clínicos integrales de
diabetes (100k filas) [conjunto de datos en Internet]. Kaggle;
2024 [Consultado el 9 de julio de 2025]. Disponible en: https://www.kaggle.com/datasets/priyamchoksi/100000-diabetes-clinical-dataset.
- Rajkomar A, Dean J, Kohane I.
Machine Learning in Medicine. N
Engl J Med.
[Internet]. 2019 [Consultado el 9 de julio de 2025];380(14):1347-58. doi: 10.1056/NEJMra1814259
- ElSayed N, Aleppo G, Aroda VR,
Bannuru RR, Brown FM, Bruemmer
D, et al. Classification and Diagnosis of Diabetes: Standards of Care in
Diabetes-2023. Diabetes Care [Internet]. 2023 [Consultado el
9 de julio de 2025];46(Suppl
1):S19-S40. doi: 10.2337/dc23-S002
- International Diabetes Federation.
Atlas de la Diabetes de la
FID [Internet]. 10ª ed. Bruselas: International Diabetes Federation;
2021. Disponible en: https://diabetesatlas.org/
- Beam AL, Kohane IS. Big
Data and Machine Learning in Health Care. JAMA. [Internet]. 2018 [Consultado el 9 de julio
de 2025];319(13):1317-1318. doi: 10.1001/jama.2017.18391
- Zhang J, Zhang Z, Zhang K, Ge X, Sun R, Zhai X. Early detection of type 2 diabetes risk:
limitations of current diagnostic criteria. Front Endocrinol (Lausanne). [Internet]. 2023 [Consultado el 9 de julio
de 2025];14:1260623. doi:
10.3389/fendo.2023.1260623
- Butalia S, Chu LM, Dover DC, Lau D, Yeung RO, Eurich DT, et al. Association Between Hemoglobin A1c
and Development of Cardiovascular Disease in Canadian Men and Women
Without Diabetes at Baseline: A Population-Based Study of 608 474 Adults. J Am Heart Assoc. [Internet]. 2024 [Consultado el 9 de julio de
2025];13(9):e031095. doi:
10.1161/JAHA.123.031095
- Lin H, Xiao N, Lin S, Liu M, Liu GG. Associations of hypertension, diabetes and heart disease risk with
body mass index in older Chinese adults: a population-based cohort study. BMJ Open [Internet]. 2024 [Consultado el 9 de
julio de 2025];14(7):e083443. doi:
10.1136/bmjopen-2023-083443
- Volpe M, Gallo g. Obesity and cardiovascular
disease: An executive document on pathophysiological and clinical links
promoted by the Italian Society of Cardiovascular Prevention (SIPREC). Front Cardiovasc Med. [Internet]. 2023 [Consultado el 9 de julio de
2025];10:1136340. doi: 10.3389/fcvm.2023.1136340
- Ahmad A, Lim LL, Morieri
ML, Tam CH, Cheng F, Chikowore T, et al.
Precision prognostics for cardiovascular disease in Type 2 diabetes: a
systematic review and meta-analysis. Commun Med (Lond). [Internet]. 2024 [Consultado el 9 de julio de
2025];4(1):11. doi: 10.1038/s43856-023-00429-z
- Bruemmer D, Singh A. Cardiometabolic
Risk: Shifting the Paradigm Toward Comprehensive Assessment JACC Adv. [Internet]. 2023 [Consultado el 9 de julio de
2025];2(18):100868. doi: 10.1016/j.jacadv.2024.100867
- Obermeyer Z, Powers B, Vogeli
C, Mullainathan S. Dissecting racial bias in an algorithm used to manage
the health of populations. Science. [Internet]. 2019 [Consultado el 9 de julio de
2025];366(6464):447-453. doi: 10.1126/science.aax2342
- Cirillo D, Catuara-Solarz S,
Morey C, Guney E, Subirats
L, Mellino S, et al. Sex and gender differences
and biases in artificial intelligence for biomedicine and healthcare. NPJ Digit Med. [Internet]. 2020 [Consultado el 9 de julio de
2025];3:81. doi: 10.1038/s41746-020-0288-5
- Chen IY, Johansson FD, Sontag D. Why Is My
Classifier Discriminatory? Adv Neural Inf Process
Syst. [Internet]. 2018 [Consultado el 9 de julio
de 2025];31. doi: 10.48550/arXiv.1805.12002
- Wiens J, Saria S, Sendak M, Ghassemi M, Liu
VX, Doshi-Velez F, et al. Do no harm: a roadmap for responsible machine
learning for health care. Nat Med. [Internet]. 2019 [Consultado el 9 de julio de
2025];25(9):1337-340. doi:110.1038/s41591-019-0548-6
- Ghassemi M, Oakden-Rayner L,
Beam AL. The false hope of current approaches to explainable artificial
intelligence in health care. Lancet Digit Health [Internet]. 2021 [Consultado el 9 de julio de
2025];3(11):e745-e750. doi:
10.1016/S2589-7500(21)00208-9
- Food and Drug Administration. Inteligencia artificial y aprendizaje
automático en software como dispositivo médico. [Internet]. Estados
Unidos: FDA; 2024 [Consultado el 9 de julio de 2025]. Disponible en: https://www.fda.gov/medical-devices/software-medical-device-samd/artificial-intelligence-and-machine-learning-software-medical-device
- The European Commission. Propuesta de Reglamento sobre un Espacio Europeo de Datos Sanitarios [Internet]. Bruselas: COM(2022) 197 final; 2022 [Consultado el 9 de julio de 2025]. Disponible en: https://health.ec.europa.eu/ehealth-digital-health-and-care/european-health-data-space
 Correo:
mlituma@hotmail.com
Correo:
mlituma@hotmail.comFuentes de financiamiento
La
investigación fue autofinanciada.
Conflictos de interés