
Editorial
Sinergia multiómica y diagnóstico clínico: hacia una medicina de
precisión y detección temprana
Multi-omic
synergy and clinical diagnosis: toward precision medicine and early detection
Heinner
Guio ¹,a
- INBIOMEDIC Research Center, Lima, Perú.
- Doctor en Ciencias Médicas.
ORCID:
https://orcid.org/0000-0003-0078-1188
Luisa
Napan-Aldazabal 1,b
- INBIOMEDIC Research Center, Lima, Perú.
- Licenciada en Biología.
ORCID:
https://orcid.org/0009-0003-7518-9565
*Autor
corresponsal:
Heinner
Guio
Correo:
heinnerguio@gmail.com
Citar como: Guio H, Napan-Aldazabal L. Sinergia
multiómica y diagnóstico clínico: hacia una medicina
de precisión y detección temprana. Rev Perú Cienc Salud. 2025;7(2): 85-8.
https://doi.org/10.37711/rpcs.2025.7.2.1
Desde
hace décadas, los análisis clínicos tradicionales (hemogramas, glucemia, perfil
lipídico, examen de orina o imágenes diagnósticas como rayos X) han sido
fundamentales para el diagnóstico de enfermedades; sin embargo, estos a menudo
detectan cambios fisiológicos o anatómicos en etapas relativamente avanzadas de
la enfermedad. En la actualidad, los avances tecnológicos nos permiten hablar
de las “ciencias ómicas”, disciplinas que estudian de
manera más completa a las moléculas, las cuales, en conjunto, componen los
mecanismos que rigen las funciones biológicas de los organismos, e integradas a
los análisis clínicos tradiciones y antecedentes familiares prometen mejorar la
detección temprana, la predicción y el diagnóstico de enfermedades (1,2).
Genómica en el diagnóstico y
predicción de enfermedades
Entre
las ciencias ómicas que podrían tener un papel más
resaltante en la optimización del diagnóstico de enfermedades, encontramos a la
genómica, que permite identificar variantes genéticas heredadas o mutaciones
somáticas relacionadas con la presencia o el riesgo de desarrollar una
enfermedad. Por ejemplo, las mutaciones en los genes BRCA1 y BRCA2 se asocian
con el incremento de riesgo de cáncer de mama y ovario, por lo que su
identificación oportuna permitiría el monitoreo y la prevención de la
enfermedad (3,4). De esta manera, la genómica complementa a la
mamografía o ecografía mamaria tradicionales al estratificar el riesgo de la
paciente y, en caso de que ya se haya desarrollado el tumor, su secuenciación
(genómica somática) revelará alteraciones genéticas que ayudan a confirmar el
diagnóstico y clasificar subtipos, más allá de lo que el microscopio muestra,
guiando terapias personalizadas.
La
diabetes tipo 2 (DT2) puede predecirse mediante factores clínicos, pero también
puede usarse la genómica para mejorar la predicción del riesgo. La DT2 tiene
una base genética que podría considerarse compleja, por lo que se deben
identificar variantes genéticas asociadas para comprender y prevenir de manera
más eficiente la enfermedad. Algunos metaanálisis
realizados en diferentes poblaciones han mostrado loci
de susceptibilidad, como: SSR1-RREB1, asociado con la regulación de glucosa en
ayunas; POU5F1-TCF19, asociado con la regulación de insulina en ayunas y
resistencia a la insulina; y ARL15, asociado al complejo mayor de
histocompatibilidad, el cual también es esencial en la respuesta inmune (6).
La hipercolesterolemia familiar (HF) también es un trastorno genético asociado a
niveles elevados de colesterol y enfermedad cardiovascular prematura. En
pacientes con sospecha de HF y con valores de lipoproteínas de baja densidad
(LDL) mayores a 190 mg/dL, se pueden realizar pruebas
genéticas de los genes LDLR, APOB o PCSK9 para confirmar el diagnóstico y
distinguir la HF de otras causas de dislipemia (6).
Se ha observado que la incorporación de la genómica aumenta el número de diagnósticos
de HF confirmados en comparación con la detección sólo en base a criterios
clínicos y analíticos (7).
Transcriptómica como
herramienta complementaria en el diagnóstico
Con
la transcriptómica se estudia el ácido ribonucleico
(ARN), como conjunto expresado en un tejido o célula, lo que aporta un análisis
de los patrones de expresión génica, como en el cáncer de mama, donde se
analiza la expresión de ciertos genes en el tejido tumoral para estimar el
riesgo de recurrencia y los efectos de la quimioterapia; todo esto, en adición
a los factores tradicionales, como tamaño tumoral, grado histológico, estado de
los ganglios y receptores hormonales, permitirá una decisión terapéutica (1,8).
Además, la transcriptómica permite la detección
temprana no invasiva mediante el análisis del ARN en sangre periférica o
“biopsia líquida”, detectando moléculas de ARN tumorales circundantes,
incluidos microRNA, iRNA y snoRNA, cuyos cambios de niveles actúan como biomarcadores precoces de varios cánceres frecuentes (mama,
colon, pulmón, hígado, páncreas, riñón, etc.) o enfermedades infecciosas
crónicas (1,9).
Proteómica y biomarcadores clínicos
La
proteómica permite medir de forma masiva las
proteínas presentes en una muestra biológica. Dado que muchas pruebas clínicas
tradicionales ya miden proteínas individuales (por ejemplo, insulina, troponina, hormonas, etc.), la proteómica
amplía el panorama, descubriendo nuevos marcadores y combinaciones proteicas
que mejoran la detección de enfermedades. Los tumores también liberan proteínas
como mecanismo de defensa hacia la respuesta inmune del individuo; estas pueden
detectarse en sangre antes de que un tumor sea visible por imágenes
radiológicas. Hoy en día, la proteómica permite el
desarrollo de paneles que identifican multiproteínas,
los cuales, integrados al análisis e identificación del ADN circulante, pueden
detectar diferentes tipos de cáncer en etapas iniciales mediante un simple
análisis de sangre (1).
Con
respecto al diagnóstico de la diabetes, la proteómica
permitiría detectar la nefropatía diabética, no sólo mediante la detección de
microalbuminuria (excreción mínima de albúmina en orina) en exámenes
tradicionales, sino también mediante la identificación de fragmentos de
colágeno y otras proteínas que se elevan años antes de que la albúmina urinaria
comience a subir. Esta información le permitiría a un nefrólogo indicar un
mejor control glucémico y tensional, antes de que ocurra un deterioro renal
clínicamente evidente (10).
Metabolómica en la
detección temprana de alteraciones metabólicas
La
metabolómica consiste en el análisis amplio de
metabolitos (azúcares, aminoácidos, ácidos grasos, etc.), que refleja el estado
metabólico global de un organismo con un enorme valor complementario para las
pruebas clínicas clásicas, especialmente para detectar desbalances metabólicos
sutiles que preceden a manifestaciones clínicas evidentes. Un ejemplo de esto
es el caso de la DT2 y otros trastornos metabólicos relacionados, donde ciertas
firmas de aminoácidos en plasma se alteran años antes de la aparición de
hiperglucemia, cuando las pruebas de glucosa o HbA1c todavía podrían estar en
un rango normal (11).
En
el caso del cáncer, tumores agresivos suelen consumir altas concentraciones de
glucosa y producir metabolitos anormales, por lo que la metabolómica
de fluidos puede complementar a las imágenes en la detección temprana, al
detectar esos cambios sutiles (1).
Por
otro lado, en el campo de las enfermedades metabólicas no oncológicas, como la
enfermedad del hígado graso no alcohólico, se han identificado perfiles de
ácidos grasos y otros metabolitos en sangre, que se correlacionan con la
presencia de inflamación hepática, mejorando la identificación de pacientes de
riesgo más allá de las pruebas tradicionales hepáticas (12).
Lipidómica y perfil
lipídico avanzado
Los
análisis tradicionales de lípidos en clínica, como colesterol total, LDL, HDL y
triglicéridos, proporcionan una visión muy limitada que puede verse enriquecida
por la lipidómica, la cual es capaz de cuantificar
diferentes especies lipídicas (fosfolípidos, esfingolípidos
como ceramidas, subtipos de ácidos grasos, etc.) que
podrían tener papeles cruciales en enfermedades cardiovasculares y metabólicas.
Se ha desarrollado un “puntaje de riesgo lipidómico”, que incorpora concentraciones de ceramidas específicas en sangre, además de los lípidos
tradicionales, para estimar el riesgo de infarto o ictus, los cuales podrían no
ser visibles con un examen de colesterol LDL. Además, al agregar estos
marcadores lipidómicos emergentes al modelo de
predicción tradicional de riesgo (que incluye colesterol, presión, tabaquismo,
etc.), la estratificación de pacientes mejora significativamente (11).
Así mismo, en el caso de la diabetes y la prediabetes se ha observado en
pacientes que, mucho antes de que la glucosa en ayunas se elevara, ya podían
detectarse cambios en ciertos fosfolípidos y triglicéridos de cadena específica
en el plasma de individuos que eventualmente se volvieron diabéticos, por lo
que un análisis lipidómico mejoraría la capacidad de
predecir el desarrollo de diabetes en los mismos (12).
Bioinformática integradora de
ciencias ómicas y datos clínicos en medicina de
precisión
En
todo este escenario, la bioinformática ocupa un lugar central en la medicina de
precisión al procesar y fusionar gran cantidad de datos procedentes de
distintas ciencias ómicas con información
tradicional, como la historia clínica electrónica, análisis de laboratorio e
imágenes, para poder proponer tratamientos personalizados (13).
Además, la integración de estos datos heterogéneos con algoritmos de
aprendizaje automático y redes neuronales facilita la extracción de patrones
complejos. Por ejemplo, existen plataformas de inteligencia artificial que han
sido diseñadas para correlacionar perfiles clínicos y genéticos de pacientes
con enfermedades autoinmunes, incorporando imágenes diagnósticas (radiografías,
tomografía, resonancias, patología digital) junto con datos genómicos y
clínicos, logrando clasificar riesgo de progresión y optimizar estrategias
terapéuticas personalizadas (13,14).
Entre algunos ejemplos concretos podemos ver que
algoritmos de inteligencia artificial aplicados a cohortes bien caracterizadas
pueden predecir la aparición de patologías o el pronóstico de un paciente, como
en el contexto de la COVID-19. El uso de secuenciación genómica de SARS-CoV-2
junto con el análisis multiómico (inmunómica,
proteómica, metabolómica)
ha permitido predecir la severidad de la enfermedad y estratificar a los
pacientes según probabilidad de desenlace grave: algo similar a los casos de prediabetes
y DT2 con metabolómica e historial clínico (15,16).
Sinergia medicina-biología
Las
perspectivas desde el punto de vista de la biología y la medicina convergen en
el campo de la medicina traslacional, por lo que un
diálogo cercano entre ambos es esencial: el médico, que atiende y diagnostica
al paciente, plantea necesidades no resueltas, mientras que el científico, que
busca entender la enfermedad a un nivel más fundamental, aporta soluciones
basadas en exploración ómica (2). Imagine
un mundo donde sólo sea necesario un marcador que detecte cierta enfermedad o
distinga subtipos que se ven iguales en radiografía y al otro lado se haya
identificado una firma proteómica que cumpla esa
función.
En
conclusión, las ciencias ómicas brindan una mirada
profunda y personalizada que complementa el panorama general que ofrecen los
análisis tradicionales, al proporcionar un diagnóstico más temprano y preciso
que enriquece la comprensión de las enfermedades y, en última instancia, mejora
los resultados clínicos (ver Figura 1). El trabajo multidisciplinario tiene un
rol fundamental en este proceso, el cual va desde un abordaje clínico hasta la
elección de nuevas innovaciones diagnósticas, incluyendo la integración de
datos multiómicos con la evaluación clínica tradicional,
lo que será cada vez más frecuente a medida que las tecnologías ómicas continúen volviéndose más precisas y accesibles.
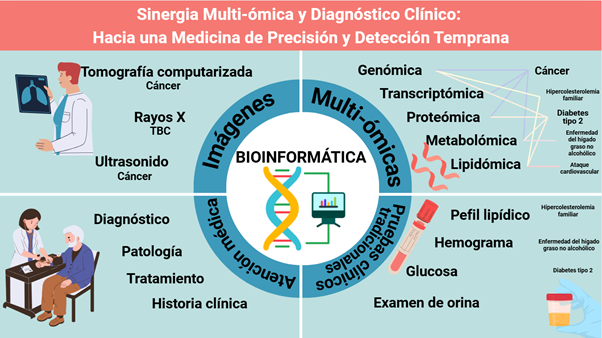
Figura 1. Sinergia multiómica
*
El análisis multiómico es la confluencia de datos
clínicos, datos de laboratorio, imágenes radiológicas y resultados de los
análisis bioinformáticos de las secuencias de ADN,
proteínas y metabolitos presentes en sangre, orina o cualquier fluido corporal.
REFERENCIAS
- Milner DA, Lennerz JK.
Technology and Future of Multi-Cancer Early Detection. Life (Basel) [Internet].
2024 [Consultado el 18 de mayo de 2025];14(7):833. https://doi.org/10.3390/life14070833
- Guio H. Towards personalized medicine: Implications
of basic sciences and the “omics” in clinical practice. Rev Peru Med Exp Salud Publica
[Internet]. 2015 [Consultado el 18 de mayo de 2025];32(4):629-632
https://doi.org/10.17843/rpmesp.2015.324.1751
- Trujillano D, Weiss M, Schneider J, Köster
J, Papachristos E, Saviouk
V, Zakharkina T, Nahavandi
N, Kovacevic L, Rolfs A. Next-Generation
Sequencing of the BRCA1 and BRCA2 Genes for the Detection of Mutations in
Breast and Ovarian Cancer Patients. J Mol Diagn [Internet]. 2015 [Consultado
el 18 de mayo de 2025];17(2):162-170. https://doi.org/10.1016/j.jmoldx.2014.10.002
- Davies H, Glodzik D, Morganella S, Yates L, Staaf
J, Zou X, et al. Detect is a predictor of BRCA1 and BRCA2 deficiency based
on mutational signatures. Nat Med. [Internet]. 2017 [Consultado el 18
de mayo de 2025];23(4):517-525. https://doi.org/10.1038/nm.4292.
- Ashenhurst J, Sazonova O, Svrchek O, Detweiler S, Kita R, Babalola L, et al. A Polygenic Score for Type 2 Diabetes Improves
Risk Stratification Beyond Current Clinical Screening Factors. Front Genet.
[Internet]. 2022 [Consultado el 18 de mayo de 2025];13:871260.
https://doi.org/10.3389/fgene.2022.871260
- Wang K, Hu T, Tai M, Shen Y, Lin S, Guo Y, et al. Pathogenicity of the LDLR c.97C>T (p.Gln33Ter) Mutation in Familial Hypercholesterolemia. Molecular genetics
& genomic medicine [Internet]. 2024
[Consultado el 18 de mayo de 2025];12(11): e70030. https://10.1002/mgg3.70030
- Hedegaard B, Bork C, Kanstrup H,
Thomsen K, Heitmann M, Bang L, et al. Genetic
testing increases the likelihood of a diagnosis of familial hypercholesterolaemia among people referred to lipid
clinics: Danish national study. Atherosclerosis [Internet]. 2023 [Consultado el 18 de mayo de
2025];373:10-16.
https://10.1016/j.atherosclerosis.2023.04.003
- Sánchez-Forgach E, Carpinteyro-Espín U, Alemán-Áviles J, Sánchez-Basurto C. Validación y aplicación
clínica de MammaPrint® en pacientes con cáncer
de mama. Cirugía y Cirujanos [Internet]. 2017 [Consultado el 18 de mayo de
2025];85(4):320-324. https://10.1016/j.circir.2016.10.019
- Guio H, Aliaga-Tobar V,
Galarza M, Pellon-Cardenas O, Capristano S, Gomez H, et al. Comparative Profiling of
Circulating Exosomal Small RNAs Derived From Peruvian Patients With Tuberculosis and Pulmonary.
Front. Cell.
Infect. Microbiol
[Internet]. 2022 [Consultado el 18 de mayo de 2025];12. https://doi.org/10.3389/fcimb.2022.909837
- Zürbig P, Jerums G, Hovind P, Macisaac RJ, Mischak H, Nielsen SE, et
al. Urinary proteomics for early
diagnosis in diabetic nephropathy. Diabetes [Internet]. 2012 [Consultado el 18 de mayo de 2025];61(12):3304-3313. https://10.2337/db12-0348
- Li J, Yu Y, Sun Y, Fu Y, Shen W, Cai L, et
al. Nuclear magnetic
resonance-based metabolomics with machine learning for predicting
progression from prediabetes to diabetes. Elife [Internet]. 2024 [Consultado el 18 de mayo de
2025];13. https://10.7554/eLife.98709
- Suvitaival T, Bondia-Pons I, Yetukuri L, Pöhö P, Nolan J, Hyötyläinen T, et al. Lipidome as a predictive tool in progression to type 2 diabetes in Finnish
men. Metab. Clin. Exp [Internet]. 2018 [Consultado el 18 de mayo de 2025];78:1-12. https://10.1016/j.metabol.2017.08.014
- Cephe A, Koçhan N, Aksel E, Ipekten F, Yerlitaş S, Zararsiz G, et
al. Bioinformatics and
biostatistics in precision medicine. Oncology: Genomics, Precision
Medicine and Therapeutic Targets [Internet]. 2023 [Consultado el 18 de mayo de 2025];189-235.
https://10.1007/978-981-99-1529-3_8
- Brancato V, Esposito G, Coppola L, Cavaliere
C, Mirabelli P, Scapicchio
C, et al. Standardizing digital biobanks: integrating imaging, genomic,
and clinical data for precision medicine. J Transl Med [Internet]. 2024 [Consultado el 18 de mayo de
2025];22(1):136. https://10.1186/s12967-024-04891-8
- Vlasova-St. Louis I, Fang D, Amer
Y, Mohei H. COVID-19-Omics Report: From
Individual Omics Approaches to Precision Medicine. Reports [Internet]. 2023 [Consultado el 18 de mayo de
2025];6(4):45. https://10.3390/reports6040045
- Song J, Wang C, Zhao T,
Zhang Y, Xing J, Zhao
X, et al. Multi-omics
approaches for biomarker discovery and precision diagnosis of prediabetes.
Front Endocrinology (Lausanne) [Internet]. 2025 [Consultado el 18 de mayo
de 2025];16:1520436. https://10.3389/fendo.2025.1520436
